
Asse 1 – Azione 1.1.4
“Sostegno alle attività collaborative di R&S per lo sviluppo di nuove tecnologie sostenibili, di nuovi prodotti e servizi”
Diagnostica in vitro di biomarcatori per la medicina personalizzata in ambito oncologico basata su sequenziamento massivo parallelo di nuova generazione
Durata del progetto: 23 mesi
Investimento: 172.366,70
Contributo: 86.183,35
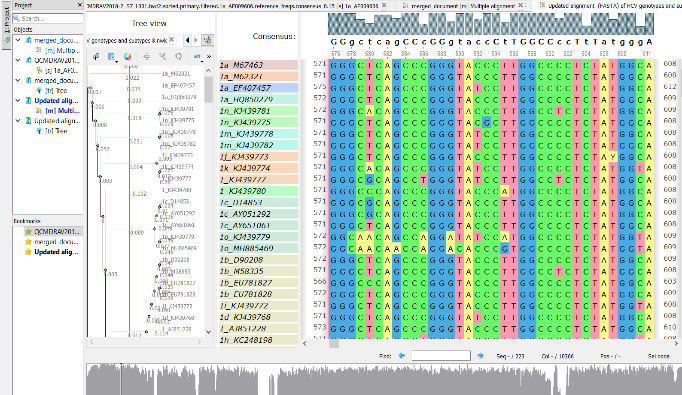
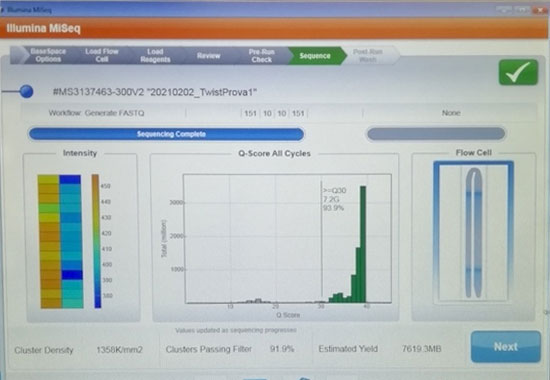
Il progetto ha come obiettivo generale lo sviluppo di approcci e metodologie di sequenziamento massivo di nuova generazione (Next Generation Sequencing), che ne consentano la diffusione economicamente sostenibile nelle Anatomie Patologiche e nei Laboratori di Diagnostica Molecolare Clinica, anche di piccole-medie dimensioni, a supporto della medicina di precisione in ambito oncologico.
Il progetto prevede lo sviluppo di Saggi NGS componibili mettendo a punto tecnologie e metodiche della fase cosiddetta library preparation, che consentano di caricare il sequenziatore con pool di librerie per target analitici diversi, massimizzando la capacità della corsa di sequenziamento e diminuendo i costi per test. Si è studiato inoltre come automatizzare i processi di progettazione e sviluppo incrementale, consentendo l’aggiunta di nuovi target in pannelli già validati.
I partner sono intervenuti nell’ automazione software dell’aggiornamento dei dati di riferimento, riducendo al minimo la manual curation nel processo di aggiornamento delle fonti (database clinici e genetici, linee guida cliniche, studi clinici, pubblicazioni scientifiche). Nel progetto ciò è stato realizzato attraverso tecniche di intelligenza artificiale.
Lo scopo finale è fornire un database di dati genici ricavati dalle pubblicazioni e forniti dagli utenti che possa essere consultato dai ricercatori. L’utilizzo dei dati presenti da parte degli utilizzatori finali viene tracciato tramite blockchain, con garanzia di integrità e inviolabilità data dalla registrazione delle operazioni sui dati. Ciò consente la protezione e la riservatezza dei dati clinici oltre a registrare il ricercatore che ha caricato i propri risultati.
Nel progetto si è proceduto anche alla standardizzazione di protocolli in biopsia liquida, considerata la nuova frontiera per la tipizzazione e stadiazione del tumore senza procedimenti invasivi per il paziente.