Descrizione
Il kit AMPLIQUALITY HCV TYPE PLUS è un IVD basato sulla tecnica Reverse Line Blot, che identifica i genotipi 1-7 del virus dell’Epatite C (HCV) ed i sottotipi a e b del genotipo 1.
Il prodotto prevede una multiplex one-step RT-PCR, a partire da RNA estratto e la successiva visualizzazione su strip degli ampliconi mediante Reverse Line Blot (RLB).
Caratteristiche rilevanti
- Per l’esecuzione del saggio sono sufficienti 10 µL di RNA estratto
- È dotato di sistema dUTP/Cod UNG per la prevenzione delle contaminazioni da carry-over
- Può essere utilizzato, nel formato per automazione, su diversi strumenti automatici per protocolli Reverse Line Blot
- Può essere corredato dal software HCV TYPE PLUS Strip Reader per l’interpretazione dei risultati
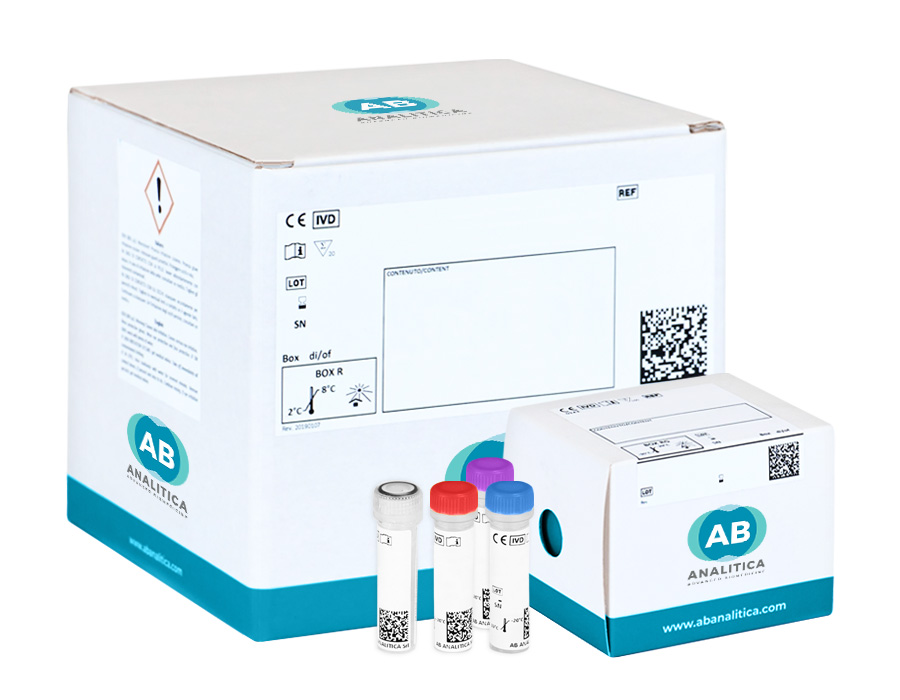
Contenuto del kit
Il kit include:
- Reagenti pronti all’uso per l’amplificazione
- Controllo positivo
- Controllo interno
- Strip e reagenti per la visualizzazione degli amplificati mediante Reverse Line Blot
- Lucido per la lettura della strip e l’interpretazione dei risultati
- Foglio archivio strip
- Vassoietti con 8 canali monouso per lo sviluppo Reverse Line Blot (solo per il formato manuale)
- CD contenente il software interpretativo HCV TYPE PLUS Strip Reader, il Manuale d’uso, la Tabella interpretativa
Approfondimento
Il virus dell’Epatite C (HCV) appartiene alla famiglia dei Flaviviridae e possiede un genoma caratterizzato da un elevato grado di variabilità. Sulla base di indagini filogenetiche e della variabilità della sequenza genomica, sono stati identificati 7 principali genotipi di HCV. I diversi genotipi differiscono nella sequenza nucleotidica per circa il 30% l’uno dall’altro. Ciascun genotipo comprende a sua volta diversi sottotipi contrassegnati dalle lettere minuscole dell’alfabeto. Ciascun sottotipo differisce a livello nucleotidico di circa il 20% rispetto ad altri membri dello stesso genotipo. A sua volta ogni sottotipo comprende numerose varianti o quasispecie dovute a mutazioni che si verificano con una certa frequenza a carico del sottotipo virale presente nel paziente stesso.
Il genoma di HCV e costituito da RNA a singolo filamento comprendente circa 9400 nucleotidi. Le regioni codificanti sono costituite dai domini del CORE, dell’Envelope e dal dominio non strutturale. La regione codificante e fiancheggiata da regioni non tradotte (UTR) altamente conservate situate in corrispondenza delle estremità 5’ e 3’.
La regione 5’UTR può fornire informazioni utili alla identificazione dei genotipi 1-5, 6a-b e 7. Tuttavia non e in grado da sola di permettere una corretta ed accurata identificazione dei sottotipi 1a e 1b, né tantomeno permette la distinzione dei sottotipi 6, non 6a e 6b, dal genotipo 1.
L’indagine simultanea delle regioni 5’UTR e CORE consente una più accurata assegnazione del genotipo e dei sottotipi virali.
Informazioni per gli ordini
| Codice | Prodotto | Confezionamento |
|---|---|---|
| 03-05-20 M | AMPLIQUALITY HCV TYPE PLUS v2.0 | 20 test |
| 03-05-20 A | AMPLIQUALITY HCV TYPE PLUS v2.0 Formato automatico | 20 test |